我认为您的错误可能来自您如何将数据包装到ggplot中,或者来自数据本身。
我没有您的数据样本,因此我使用了样本数据库Toothgrowth和您的代码stat_compare_mean,得到了您想要的显示。
这是我的代码:
library(ggpubr)
data("ToothGrowth")
# Box plot faceted by "dose"
p <- ggboxplot(ToothGrowth,x = "supp",y = "len",color = "supp",palette = "jco",add = "jitter",facet.by = "dose",short.panel.labs = FALSE)
# Adding stat_compare_means
p + stat_compare_means(show.legend=FALSE,label.x.npc = 0.5,label.y.npc = 0.93,color = "black",size = 4) + theme_bw()
这是情节:

如果您改用它,则绘图效果更好:
p + stat_compare_means() + theme_bw()

更新:提示以显示最终情节
因此,我尝试重现您的数据,以重现绘制得出的错误,并且我成功使用本文R: ggplot2 - Kruskal-Wallis test per facet
中描述的技巧来绘制p值
这是我用来模仿您的数据的代码:
set.seed(1)
# defining the sample dataset AJCC
PSA_levels <- rnorm(100,mean = 2,sd = 2)
AJCC_data <- data.frame(cbind(PSA_levels))
x <- NULL
for(i in 1:100) {x <- c(x,sample(1:4,1))}
AJCC_data$score <- x
AJCC_data$Method <- 'AJCC'
# defining the sample dataset Gleason
PSA_levels <- rnorm(100,mean = 2.5,sd = 1)
Gleason_data <- data.frame(cbind(PSA_levels))
x <- NULL
for(i in 1:100) {x <- c(x,sample(5:10,1))}
Gleason_data$score <- x
Gleason_data$Method <- 'Gleason'
# defining the sample dataset TNM
PSA_levels <- rnorm(100,sd = 1)
TNM_data <- data.frame(cbind(PSA_levels))
x <- NULL
for(i in 1:100) {x <- c(x,sample(1:30,1))}
TNM_data$score <- x
TNM_data$Method <- 'TNM'
df <- rbind(AJCC_data,Gleason_data,TNM_data)
df$score <- as.factor(df$score)
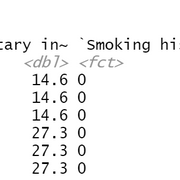
这里是df的输出,看起来与您的数据tabcourt
类似
> str(df)
'data.frame': 300 obs. of 3 variables:
$ PSA_levels: num 0.747 2.367 0.329 5.191 2.659 ...
$ score : Factor w/ 30 levels "1","2","3","4",..: 2 1 2 2 2 3 1 2 3 3 ...
$ Method : chr "AJCC" "AJCC" "AJCC" "AJCC" ...
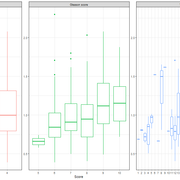
然后,我尝试重现刻面的箱线图:
library(ggplot2)
library(ggpubr)
g <- ggplot(df,aes(x = score,y = PSA_levels,color = Method))
p <- g + facet_wrap(.~Method,scales = 'free_x')
p <- p + geom_boxplot()
p <- p + theme_bw()
当我尝试使用stat_compare_means函数在图形上添加p值时,出现了与您相同的绘制错误。因此,根据上面引用的帖子,我使用了软件包dplyr为每个组生成Kruskal Wallis检验的pvalue。
library(dplyr)
ptest <- df %>% group_by(Method) %>% summarize(p.value = kruskal.test(PSA_levels ~score)$p.value)
这是ptest的输出:
> ptest
# A tibble: 3 x 2
Method p.value
<chr> <dbl>
1 AJCC 0.575
2 Gleason 0.216
3 TNM 0.226
现在,我可以通过执行以下操作添加该箱线图:
p + geom_text(data = ptest,aes(x = c(2,3,10),y = c(6,6,6),label = paste0("Kruskal-Wallis\n p=",round(p.value,3))))
在这里,您得到的是:

因此,我认为这是因为stat_compare_means无法理解要比较的组以及如何在图形上表示所有统计比较。在ggplot之外进行测试,然后添加为geom_text自变量可以解决这种情况。
希望它可以处理您的真实数据!
,
谢谢您的解决方法!!!它确实有效,但是我必须添加:+scale_x_discrete()否则我会得到Error: Discrete value supplied to continuous scale
如果别人遇到这种情况,这就是我使用的代码:
ptest = tabcourt %>% group_by(Method) %>%summarize(p.value=kruskal.test("mtDNA copy number"~Score)$p.value)
p2 = ggplotly(ggplot(subset(tabcourt,!is.na(Score) & !is.na("mtDNA copy number")),aes(x =Score,y ="mtDNA copy number",color=Method))
+ scale_x_discrete()
+ geom_text(data = ptest,aes(x =c(2,y= c(1.5,1.5,1.5),3))))
+ facet_grid(.~Method,scales='free')
+ geom_boxplot()
+ theme_bw())
奇怪的是stat_compare_means很难做到!
本文链接:https://www.f2er.com/3141111.html